AIDプロジェクト
メンバー
2023/06/02
 |
ナシム ベガム | 特定准教授 |
|---|---|---|
 |
小林 牧 | 特定准教授 |
 |
Santosh Gothwal | 研究員 |
 |
I Ketut Gunarta | 研究員 |
 |
Wang Wencong | 博士課程学生 |
 |
Chen Xi | 博士課程学生 |
 |
Maidaiti Baerlike | 研究生 |
 |
Ahmed Refaat | 研究生 |
 |
中田 幹代 | テクニシャン |
 |
松井 寿美子 | テクニシャン |
 |
Tabassum Ara | テクニシャン |
[抗体研究の歩み]
ワクチンの開発は医学研究による人類の健康への最大の貢献である。初めてのワクチンは1798年Jennerによって試みられた天然痘ワクチンである。 その後100年以上に渡って何故ワクチンが感染症を予防することが出来るのかは謎であった。この謎が解けたのは抗体の存在が明らかになったからである。 Boehingと北里はJennerから100年後にジフテリア毒素の免疫により血清中に中和活性を持つ物質が現れることを発見した。 これが抗体の存在を示した初めての報告である。やがて抗体分子の構造が明らかになり,そして抗原刺激によって最初にIgMが誘導され, 遅れてIgGの産生誘導が見られることが明らかになった。 2回目に同じ抗原を投与すると最初から抗原と強い結合能力を持ったIgG抗体が産生される。すなわちこれがワクチンの原理であり, 生体が抗原投与による免疫記憶によって抗原排除に有利な抗体を作るという現象である。免疫抗体記憶の本質は「IgMからIgGへクラススイッチ」 と「体細胞突然変異」による抗原との結合能力の上昇である。その後の研究によりこの2つの現象はいずれも「DNAの組換え」と「配列変異」として ゲノム上に刻印されることが明らかとなった。
AIDはその構造や細胞生物学的特徴からRNA編集酵素である可能性があり、そのCSR誘導の分子機構として未知の標的RNAを編集し、 その結果CSR やSHMを起こす新たな組換え酵素が作られるという作業仮説を立て研究を進めている。もしその予想が真実であるならば、 抗体の多様性は RNA 情報の操作を介してゲノム情報を変換するシステムにより生み出されると言えるだろう。
[AIDの発見まで]
それでは何が抗体記憶をゲノムに刻印するのだろうか。当研究室は抗体クラススイッチの分子機構の解明に一貫して取り組み, この分野において常に世界をリードする研究を発表してきた。1978年に抗体遺伝子のDNAレベル での組換え(クラススイッチ組換え:CSR) がクラススイッチ分子的基盤であることを提唱し, そのモデルを実際に証明した。そしてBoehringと北里から100年後にあたる1999年, 当研究室にてCSRに必須のリンパ球特異的な因子Activation induced cytidine deaminase (AID)が発見され, 2000年にこの分子がCSRに必須 である事が示された。AID が欠損するとCSRに加えて, 抗体可変領域遺伝子の体細胞突然変異(somatic hypermutation: SHM)までもが全く起こらなくなる事を明らかにした。 それまで, クラススイッチは比較的長い遺伝子領域の欠失と組換であるのに対してSHMは遺伝子変異であったため, 2つの一見異なる現象が同じAID という分子によって制御されていること, すなわちAIDが抗体記憶をゲノムに刻印することは当時, 驚きに値することだった。更に, 繊維芽細胞等 Bリンパ球以外の細胞にAIDを強制発現させると, 通常は活性化B細胞にしか起こらない筈のCSRやSHM活性を誘導できることを発見した。 またBリンパ球の腫瘍化に関連する免疫グロブリン遺伝子とc-myc遺伝子との相互転座においてもAIDが引き金となることに加え, 各種慢性感染症により 異常発現したAIDによるDNA改変誘導の発がんへの関与が示唆され, 免疫学の領域を超えた他の研究領域への展開も見せ始めている。
[AID研究の発展 -2004~2011-AIDドメイン解析]
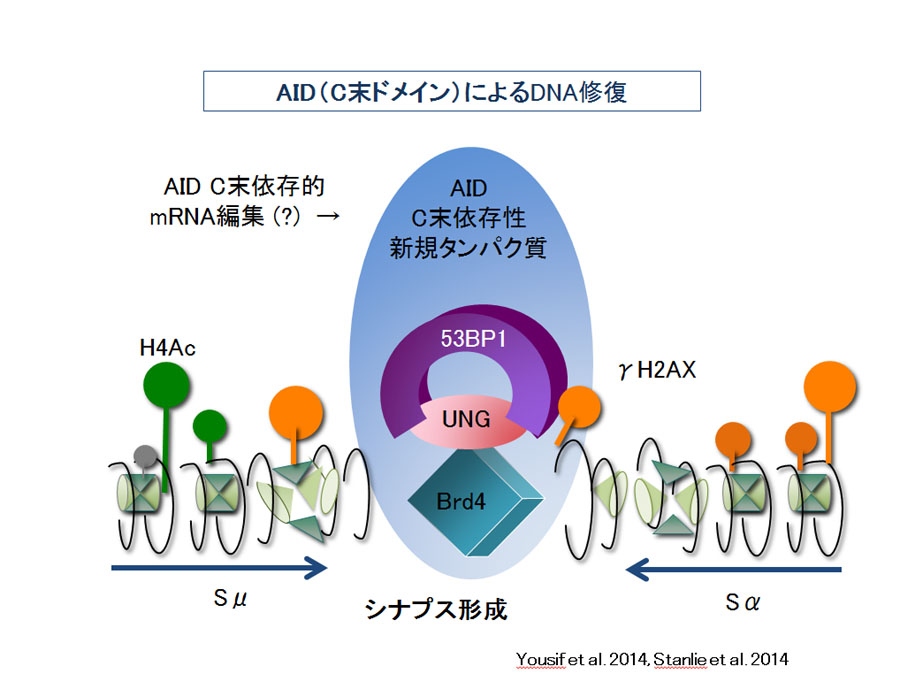
当研究室では, AIDがSHMとCSRにおいて果たす分子メカニズムについて研究を続行している。まず, AIDは N末端ドメインと中央の シチジンデミナーゼドメインに依存する「一本鎖DNA切断」(CSR とSHM の両者に必要)機能を持ち、またC末端ドメインに依存する「組換え」 (CSRにのみ必要)機能を持つことを, 各種のAID 変異体を用いて明らかにした。N末端ドメインやシチジンデミナーゼドメインのAID変異体はDNA 切断活性を持たず、CSRもSHMも起こさない。一方、C末端ドメインに変異や欠失を持つAID変異体はDNA切断活性を持つものの、 組換え活性がないため、CSRを起こさなかった。このドメイン構造の認識はAIDのDNA脱アミノ化仮説に疑問を投じる根拠となり、 また後のAIDの二量体化解析の上で非常に重要な手がかりとなった。[AID研究の発展 -2008~2011- AID依存性DNA切断におけるTop1、ヒストン修飾の重要性]
AIDはシチジン(C)脱アミノ活性を有し, RNA 編集酵素APOBEC1 との構造類似性からRNA 編集酵素であろうと推測される。つまりDNAを直接切断する 活性が無いため, AID誘導性にDNAを切断する酵素を探索した結果, 2009 年, AID によるDNA 切断がTopoisomerase 1 (Top1) による一本鎖切断から開始 されることを示した。Top1は普遍的にDNA上に分布しDNA二重鎖構造の維持に活躍する酵素であるにも関わらず, AIDに制御されるTop1による切断は なぜ免疫グロブリン遺伝子などの限定された領域にのみ起きるのか。これは「転写」と, AIDにより切断される「DNA配列の特異性 (=反復配列に富んでいること)」, さらにDNA切断部位に「特異的なヒストン修飾」が集積することの3つから説明することができる。 すなわち、DNA上の「転写」によるDNA二重鎖の緩みに続き、 AIDの活性化によりTop1タンパク質が減少し, 「反復配列」に起こるNon-B 構造の形成が 増強されるため, Top1自身による一本鎖切断後にDNA 構造の制約のため回転できず, DNAが切断されたまま修復されない状況が起きる。 また, AIDによる免疫グロブリン遺伝子座の切断には, SSRP1とSPT16からなるFACT複合体の蓄積が必須であること, FACT複合体の機能はH3K4me3の ヒストン修飾に必要なことを明らかにした。[AIDのターゲットはDNAかRNAなのか? -2009~2014- UNGやAPE1の機能解明]
当研究室による2000年のAID報告に続き, 2002 年, E.coli 中やin vitroでのDNA のAID によるC 脱アミノ活性が検出されることからNeuburger 等によりDNA 脱アミノ仮説が提唱された。この説ではAID はDNA 上のCを脱アミノしウリジン(U)を形成し, 生じたU/G(グアニン)ミスマッチを UNGとAPE1の塩基除去修復経路の酵素群が処理する結果, DNA切断が起こるとされた。現在もこの論争は続いているが, 我々の最近の研究によりUNGと APE1は, いずれもDNA切断に関わらずにSHMには不要で, むしろ, 両者はDNA切断後の組換え過程において必要であることを証明した。 つまり, AIDによるDNA切断はUNGやAPE1によっては説明不可能である。また, 仮にAIDがDNAを脱アミノ化するだけならば、 C末端とN末端が全く異なる機能を持つことが説明できない。DNA切断モデルを支持する追加の証拠は得られておらず, むしろ, 典型的なRNA 編集酵素APOBEC1 ですらE. coli 中のDNA 脱アミノ反応を起こすことが示され, in vitroやE.coli中でのDNAの脱アミノ化現象が必ずしも in vivoのDNA脱アミノ化を証明しているわけではないと考えられる。[多方面からのAIDの分子機能解明 -2012~2015- miRNAを介したAIDによるTop1制御, AID共役因子の同定, AIDの単量体と二量体形成との機能相関, Top1のクロマチン複合体への集積]
AIDの活性化によりTop1が減少することが明らかになったがその分子メカニズムについてはどのように考えられるだろうか? AID依存性のDNA切断には 新規のタンパク質合成は必要ないことがわかっていることから、AIDはタンパク質でなく、RNAの変化を制御することが予想される。 AID がmiRNAの前駆体RNAを編集してTop1 3’UTRに結合するmiRNAの上昇を招くのではないだろうか。それによりTop1 mRNA の翻訳を抑制する。 Top1 が低下するため転写が活発な反復配列領域でヘリックスの緩みによるNon-B 構造形成が促進されるが, ヘリックスを修正しようとするTop1 がDNA に結合したあと不可逆的に結合したままでDNA 切断を生じるであろう。この期間に, AIDによるTop1のタンパク質減少はTop1 3’UTRの特定部位 へのmiRNA-Ago2複合体の集積が引き起こされることを明らかにした。また、典型的なRNA脱アミノ酵素APOBEC1は共役因子ACFと複合体になって初めて編集ターゲットであるAPOB100mRNAを結合することが可能となり, 脱アミノ化反応を起こす。演繹的にAIDは単独ではRNAに結合できないと考えられ, CSRやSHMに特異的なAID共役因子は何かということが長年の課題 であった。APOBEC1の共役因子ACFがheterogenous nuclear RNA binding protein (hnRNP)ファミリーのRNA結合タンパク質であることから, 各種hnRNPファミリーのタンパク質のノックダウンをクラススイッチモデル系でテストすることにより, 最終的にDNA切断に特異的なhnRNPKとCSRに 特異的なhnRNPLを同定した。AIDの2つの機能特異的な共役因子が同定されたことで、AIDのターゲットRNAにつながる知見である。 また、AIDは二量体を形成することが予想されていたが、単量体でDNA切断をおこすのに対し、CSR(DNA修復)にはAIDの二量体形成が必要であることが 明らかになった。
Top1の複合体形成を調べた結果, Top1がSmarca4を介して免疫グロブリンS領域にリクルートされること、FACT複合体がTop1とH3K4me3とを結合する アダプターとして働くことが明らかになり、Top1のS領域局所での分子動態がより明らかになった。
[2015~ AIDのRNA編集ターゲットの同定とその機能, またAIDの構造と機能相関解明に向かって]
以上をふまえ, 我々は, 抗体遺伝子におけるAIDによるCSR 及びSHM誘導の分子機構の解明を目標に研究を進めている。 AIDにより、複数のRNAターゲット分子が編集され、miRNA前駆体についてはTop1その他のタンパク質分子の翻訳抑制を起こし最終的にはDNA切断に、 mRNA前駆体については新規のタンパク質を合成させることからDNA切断後の修復/組換えを完了させるものであると考えられる。 もしその予想が真実であるならば, 抗体の多様性は RNA 情報の操作を介してゲノム情報を変換するシステムにより生み出されると言えるだろう。[代表的な論文]
1. Activation-induced cytidine deaminase (AID) deficiency causes the autosomal recessive form of the hyper-IgM syndrome (HIGM2).Revy, P., Muto, T., Levy, Y., Geissmann, F., Plebani, A., Sanal, O., Catalan, N., Forveille, M., D.-Lagelouse, R., Gennery, A., Tezcan, I., Ersoy, F., Kayserili, H., Ugazio, A.G., Brousse, N., Muramatsu, M., Notarangelo, L.D., Kinoshita, K., Honjo, T., Fischer, A. and Durandy, A.
Cell 102 565-575 (2000) [PubMed]
2. In situ class switching and differentiation to IgA-producing cells in the gut lamina propria.
Fagarasan, S., Kinoshita, K., Muramatsu, M., Ikuta, K. and Honjo, T.
Nature 413 639-643 (2001) [PubMed]
3. Constitutive expression of AID leads to tumorigenesis.
Okazaki, I., Hiai, H., Kakazu, N., Yamada, S., Muramatsu, M., Kinoshita, K. and Honjo, T.
J. Exp. Med. 197 1173-1181 (2003) [PubMed]
4. AID mutant analyses indicate requirement for class-switch-specific cofactors.
Ta, V-T., Nagaoka, H., Catalan, N., Durandy, A., Fischer, A., Imai, K., Nonoyama, S., Tashiro, J., Ikegawa, M., Ito, S., Kinoshita, K., Muramatsu, M. and Honjo, T.
Nature Immunol. 4 843-848 (2003) [PubMed]
5. Uracil DNA glycosylase activity is dispensable for immunoglobulin class switch.
Begum, N. A., Kinoshita, K., Kakazu, N., Muramatsu, M., Nagaoka, H., Shinkura, R., Biniszkiewicz, D., Boyer, L. A., Jaenisch, R. and Honjo, T.
Science 305 1160-1163 (2004) [PubMed]
6. AID-induced decrease in topoisomerase 1 induces DNA structural alteration and DNA cleavage for class switch recombination.
Kobayashi M, Aida M, Nagaoka H, Begum NA, Kitawaki Y, Nakata M, Stanlie A, Doi T, Kato L, Okazaki IM, Shinkura R, Muramatsu M, Kinoshita K, Honjo T.
Proc Natl Acad Sci U S A. 106:22375-80 (2009) [PubMed]
7. Histone3 lysine4 trimethylation regulated by the facilitates chromatin transcription complex is critical for DNA cleavage in class switch recombination.
Stanlie A, Aida M, Muramatsu M, Honjo T, Begum NA.
Proc Natl Acad Sci U S A. 107:22190-5 (2010) [PubMed]
8. Mice carrying a knock-in mutation of Aicda resulting in a defect in somatic hypermutation have impaired gut homeostasis and compromised mucosal defense.
Wei M, Shinkura R, Doi Y, Maruya M, Fagarasan S, Honjo T.
Nat Immunol. 12:264-70 (2011) [PubMed]
9. The AID dilemma: infection, or cancer?
Honjo T, Kobayashi M, Begum N, Kotani A, Sabouri S, Nagaoka H.
Adv Cancer Res. 113:1-44 (2012) [PubMed]
10. Nonimmunoglobulin target loci of activation-induced cytidine deaminase (AID) share unique features with immunoglobulin genes.
Kato L, Begum NA, Burroughs AM, Doi T, Kawai J, Daub CO, Kawaguchi T, Matsuda F, Hayashizaki Y, Honjo T.
Proc Natl Acad Sci U S A. 109 :2479-84 (2012) [PubMed]
11. Chromatin reader Brd4 functions in Ig class switching as a repair complex adaptor of nonhomologous end-joining.
Stanlie A, Yousif AS, Akiyama H, Honjo T, Begum NA.
Mol Cell 55:97-110 (2014) [PubMed]
12. APE1 is dispensable for S-region cleavage but required for its repair in class switch recombination.
Xu, J., Husain, A., Hu, W., Honjo, T. and Kobayashi, M.
Proc. Natl. Acad. Sci. USA 111 17242-17247 (2014) [PubMed]
13. Identification of DNA cleavage- and recombination-specific hnRNP cofactors for activation-induced cytidine deaminase.
Hu W, Begum NA, Mondal S, Stanlie A, Honjo T.
Proc Natl Acad Sci U S A. 112:5791-6 (2015) [PubMed]